一、Meningkatkan sensitivitas sistem reaksi:
1. Isolasi RNA berkualitas tinggi:
Sintesis cDNA yang sukses berasal dari RNA berkualitas tinggi.RNA berkualitas tinggi setidaknya harus panjang penuh dan bebas dari penghambat transkriptase balik seperti EDTA atau SDS.Kualitas RNA menentukan jumlah maksimum informasi sekuens yang dapat Anda transkripsikan ke dalam cDNA.Metode pemurnian RNA yang umum adalah metode satu langkah menggunakan guanidine isothiocyanate/acid phenol.Untuk mencegah kontaminasi oleh RNase dalam jumlah kecil, RNA yang diisolasi dari sampel kaya RNase (seperti pankreas) perlu disimpan dalam formaldehida untuk mempertahankan RNA berkualitas tinggi, terutama untuk penyimpanan jangka panjang.RNA yang diekstraksi dari hati tikus pada dasarnya terdegradasi setelah disimpan dalam air selama satu minggu, sedangkan RNA yang diekstraksi dari limpa tikus tetap stabil setelah disimpan dalam air selama 3 tahun.Selain itu, transkrip yang lebih panjang dari 4 kb lebih sensitif terhadap degradasi oleh RNase jejak daripada transkrip kecil.Untuk meningkatkan stabilitas penyimpanan sampel RNA, RNA dapat dilarutkan dalam formamida deionisasi dan disimpan pada -70°C.Formamida yang digunakan untuk mengawetkan RNA harus bebas dari puing-puing yang merusak RNA.RNA dari pankreas dapat disimpan dalam formamida setidaknya selama satu tahun.Saat mempersiapkan untuk menggunakan RNA, Anda dapat menggunakan metode berikut untuk mengendapkan RNA: tambahkan NaCl ke 0,2M dan 4 kali volume etanol, tempatkan pada suhu kamar selama 3-5 menit, dan sentrifugasi pada 10.000×g selama 5 menit.
2. Gunakan transkriptase balik RNaseH-inaktif (RNaseH-):
Inhibitor RNase sering ditambahkan untuk membalikkan reaksi transkripsi untuk meningkatkan panjang dan hasil sintesis cDNA.Inhibitor RNase harus ditambahkan selama reaksi sintesis untai pertama dengan adanya buffer dan agen pereduksi (seperti DTT), karena proses sebelum sintesis cDNA mendenaturasi inhibitor, sehingga melepaskan RNase terikat yang dapat menurunkan RNA.Inhibitor protein RNase hanya mencegah degradasi RNA oleh RNase A, B, C, dan tidak mencegah RNase pada kulit, jadi berhati-hatilah agar tidak memasukkan RNase dari jari Anda meskipun menggunakan inhibitor ini.
Reverse transcriptase mengkatalisis konversi RNA menjadi cDNA.Baik M-MLV dan AMV memiliki aktivitas RNaseH endogen selain aktivitas polimerase mereka sendiri.Aktivitas RNaseH dan aktivitas polimerase bersaing satu sama lain untuk untai hibrida yang terbentuk antara templat RNA dan primer DNA atau untai ekstensi cDNA, dan menurunkan untai RNA dalam kompleks RNA:DNA.Template RNA yang terdegradasi oleh aktivitas RNaseH tidak dapat lagi berfungsi sebagai substrat yang efektif untuk sintesis cDNA, yang mengurangi hasil dan panjang sintesis cDNA.Oleh karena itu, akan bermanfaat untuk menghilangkan atau sangat mengurangi aktivitas RNaseH dari reverse transcriptase.。
SuperScript Ⅱ reverse transcriptase, RNaseH- MMLV reverse transcriptase dan thermoScript reverse transcriptase, RNaseH- AMV, dapat memperoleh lebih banyak cDNA full-length daripada MMLV dan AMV.Sensitivitas RT-PCR akan dipengaruhi oleh jumlah sintesis cDNA.ThermoScript jauh lebih sensitif daripada AMV.Ukuran produk RT-PCR dibatasi oleh kemampuan reverse transcriptase untuk mensintesis cDNA, terutama saat mengkloning cDNA yang lebih besar.Dibandingkan dengan MMLV, SuperScripⅡ secara signifikan meningkatkan hasil produk RT-PCR yang panjang.RNaseH-reverse transcriptase juga telah meningkatkan termostabilitas, sehingga reaksi dapat dilakukan pada suhu yang lebih tinggi dari suhu normal 37-42°C.Di bawah kondisi sintesis yang disarankan, gunakan oligo(dT) primer dan 10 μCi dari [α-P]dCTP.Hasil total untai pertama dihitung dengan menggunakan metode presipitasi TCA.CDNA panjang penuh dianalisis menggunakan pita yang disortir ukuran yang dipotong dan dihitung pada gel agarosa alkali.
3. Naikkan suhu inkubasi untuk transkripsi terbalik:
Suhu inkubasi yang lebih tinggi membantu membuka struktur sekunder RNA, meningkatkan hasil reaksi.Untuk sebagian besar cetakan RNA, menginkubasi RNA dan primer pada suhu 65°C tanpa buffer atau garam, diikuti dengan pendinginan cepat di atas es akan menghilangkan sebagian besar struktur sekunder dan memungkinkan primer untuk berikatan.Namun, beberapa cetakan masih memiliki struktur sekunder, bahkan setelah denaturasi panas.Amplifikasi dari template yang sulit ini dapat dilakukan dengan menggunakan ThermoScript Reverse Transcriptase dan menempatkan reaksi transkripsi balik pada suhu yang lebih tinggi untuk meningkatkan amplifikasi.Suhu inkubasi yang lebih tinggi juga dapat meningkatkan spesifisitas, terutama ketika primer spesifik gen (GSP) digunakan untuk sintesis cDNA (lihat Bab 3).Jika menggunakan GSP, pastikan Tm primer sama dengan suhu inkubasi yang diharapkan.Jangan gunakan oligo(dT) dan primer acak di atas 60°C.Primer acak membutuhkan inkubasi pada 25°C selama 10 menit sebelum meningkat menjadi 60°C.Selain menggunakan suhu transkripsi balik yang lebih tinggi, spesifisitas juga dapat ditingkatkan dengan secara langsung mentransfer campuran RNA/primer dari suhu denaturasi 65°C ke suhu inkubasi transkripsi balik dan menambahkan campuran reaksi 2x yang telah dihangatkan sebelumnya (sintesis cDNA hot-start).Pendekatan ini membantu mencegah pasangan basa antarmolekul yang terjadi pada suhu yang lebih rendah.Pengalihan temperatur ganda yang diperlukan untuk RT-PCR dapat disederhanakan dengan menggunakan thermal cycler.
Polimerase termostabil T bertindak sebagai DNA polimerase dengan adanya Mg2+ dan sebagai RNA polimerase dengan adanya Mn2+.Itu dapat tetap hangat pada suhu maksimum 65 ° C.Namun, keberadaan Mn2+ selama PCR mengurangi fidelitas, yang membuat polimerase Tth kurang cocok untuk amplifikasi presisi tinggi, seperti kloning cDNA.Selain itu, Tth memiliki efisiensi transkripsi balik yang rendah, yang mengurangi sensitivitas, dan, karena transkripsi balik dan PCR dapat dilakukan dengan enzim tunggal, reaksi kontrol tanpa transkripsi balik tidak dapat digunakan untuk membandingkan produk amplifikasi cDNA dengan DNA genom yang terkontaminasi.Produk amplifikasi dipisahkan.
4. Aditif yang mempromosikan transkripsi terbalik:
Aditif termasuk gliserol dan DMSO ditambahkan ke reaksi sintesis untai pertama, yang dapat mengurangi stabilitas untai ganda asam nukleat dan melepaskan struktur sekunder RNA.Hingga 20% gliserol atau 10% DMSO dapat ditambahkan tanpa memengaruhi aktivitas SuperScript II atau MMLV.AMV juga dapat mentolerir hingga 20% gliserol tanpa kehilangan aktivitas.Untuk memaksimalkan sensitivitas RT-PCR dalam reaksi transkripsi balik SuperScriptⅡ, gliserol 10% dapat ditambahkan dan diinkubasi pada suhu 45°C.Jika 1/10 produk reaksi transkripsi balik ditambahkan ke PCR, maka konsentrasi gliserol dalam reaksi amplifikasi adalah 0,4%, yang tidak cukup untuk menghambat PCR.
5. Pengobatan RNaseH:
Pengobatan reaksi sintesis cDNA dengan RNaseH sebelum PCR dapat meningkatkan sensitivitas.Untuk beberapa templat, diperkirakan bahwa RNA dalam reaksi sintesis cDNA mencegah pengikatan produk amplifikasi, dalam hal ini pengobatan RNaseH dapat meningkatkan sensitivitas.Secara umum, perawatan RNaseH diperlukan saat memperkuat templat target cDNA panjang penuh yang lebih panjang, seperti scherosis tuberous II salinan rendah.Untuk templat yang sulit ini, perawatan RNaseH meningkatkan sinyal yang dihasilkan oleh SuperScript II atau cDNA yang disintesis AMV.Untuk sebagian besar reaksi RT-PCR, pengobatan RNaseH bersifat opsional, karena langkah denaturasi PCR pada 95°C umumnya menghidrolisis RNA dalam kompleks RNA:DNA.
6. Peningkatan Metode Deteksi RNA Kecil:
RT-PCR sangat menantang ketika hanya sejumlah kecil RNA yang tersedia.Glikogen ditambahkan sebagai pembawa selama isolasi RNA membantu meningkatkan hasil sampel kecil.Glikogen bebas RNase dapat ditambahkan bersamaan dengan penambahan Trizol.Glikogen larut dalam air dan dapat disimpan dalam fase berair dengan RNA untuk membantu pengendapan selanjutnya.Untuk sampel kurang dari 50 mg jaringan atau 106 sel kultur, konsentrasi glikogen bebas RNase yang direkomendasikan adalah 250 μg/ml.
Menambahkan BSA asetat ke reaksi transkripsi terbalik menggunakan SuperScript II dapat meningkatkan sensitivitas, dan untuk sejumlah kecil RNA, mengurangi jumlah SuperScript II dan menambahkan 40 unit RNaseOut nuclease inhibitor dapat meningkatkan tingkat deteksi.Jika glikogen digunakan dalam proses isolasi RNA, masih disarankan untuk menambahkan BSA atau RNase inhibitor saat menggunakan SuperScript II untuk reaksi transkripsi balik.
二、Meningkatkan spesifisitas RT-PCR
1. Sintesis CND:
Sintesis cDNA untai pertama dapat dimulai dengan menggunakan tiga metode berbeda, spesifisitas relatif yang memengaruhi jumlah dan jenis cDNA yang disintesis.
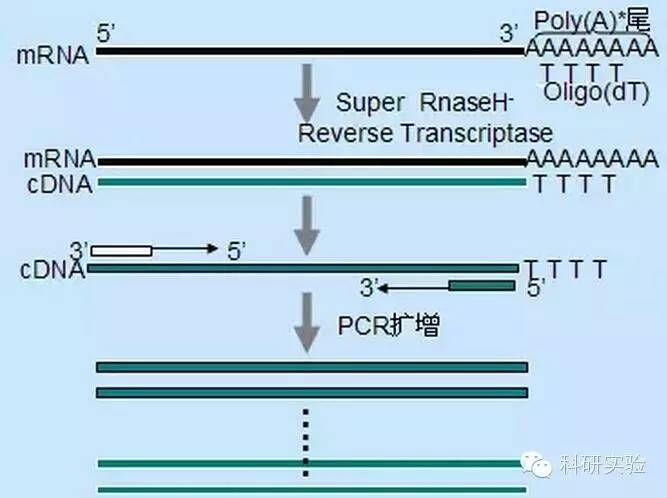
Metode primer acak adalah yang paling tidak spesifik dari ketiga metode tersebut.Primer anil di beberapa situs di seluruh transkrip, menghasilkan cDNA pendek dan sebagian panjang.Metode ini sering digunakan untuk mendapatkan urutan ujung 5' dan untuk mendapatkan cDNA dari cetakan RNA dengan daerah struktur sekunder atau dengan situs terminasi yang tidak dapat direplikasi oleh reverse transcriptase.Untuk mendapatkan cDNA terpanjang, rasio primer terhadap RNA pada setiap sampel RNA perlu ditentukan secara empiris.Konsentrasi awal primer acak berkisar antara 50 hingga 250 ng per 20 μl reaksi.Karena cDNA yang disintesis dari RNA total menggunakan primer acak terutama adalah RNA ribosom, poli(A)+RNA umumnya dipilih sebagai templat.
Oligo(dT) primer lebih spesifik daripada primer acak.Ia berhibridisasi ke ekor poli(A) yang ditemukan pada ujung 3′ dari sebagian besar mRNA eukariotik.Karena poli(A)+ RNA kira-kira 1% sampai 2% dari total RNA, jumlah dan kompleksitas cDNA jauh lebih sedikit dibandingkan dengan primer acak.Karena spesifisitasnya yang tinggi, oligo(dT) umumnya tidak memerlukan optimalisasi rasio RNA terhadap primer dan pemilihan poli(A)+.Disarankan untuk menggunakan 0,5μg oligo(dT) per 20μl sistem reaksi.oligo(dT)12-18 cocok untuk sebagian besar RT-PCR.Sistem ThermoScript RT-PCR menawarkan oligo(dT)20 karena stabilitas termalnya yang lebih baik untuk suhu inkubasi yang lebih tinggi.
Primer spesifik gen (GSP) adalah primer paling spesifik untuk langkah transkripsi terbalik.GSP adalah oligonukleotida antisense yang secara khusus dapat berhibridisasi ke urutan target RNA, tidak seperti primer acak atau oligo(dT), yang dianil ke semua RNA.Aturan yang sama yang digunakan untuk mendesain primer PCR berlaku untuk desain GSP dalam reaksi transkripsi terbalik.GSP dapat menjadi urutan yang sama dengan primer amplifikasi yang dianil ke ujung mRNA paling 3′, atau GSP dapat dirancang untuk anil di hilir primer amplifikasi terbalik.Untuk beberapa subjek yang diamplifikasi, lebih dari satu primer antisense perlu dirancang untuk keberhasilan RT-PCR karena struktur sekunder RNA target dapat mencegah pengikatan primer.Direkomendasikan untuk menggunakan 1 pmol antisense GSP dalam 20 μl reaksi sintesis untai pertama.
2. Naikkan suhu inkubasi untuk transkripsi terbalik:
Untuk mengambil keuntungan penuh dari keuntungan penuh dari spesifisitas GSP, reverse transcriptase dengan termostabilitas yang lebih tinggi harus digunakan.Reverse transcriptase termostabil dapat diinkubasi pada suhu yang lebih tinggi untuk meningkatkan kekakuan reaksi.Misalnya, jika GSP dianil pada suhu 55°C, spesifisitas GSP tidak akan digunakan sepenuhnya jika AMV atau M-MLV digunakan untuk transkripsi balik pada tingkat keketatan rendah 37°C.Namun, SuperScript II dan ThermoScript dapat direaksikan pada suhu 50°C atau lebih tinggi, yang akan menghilangkan produk non-spesifik yang dihasilkan pada suhu lebih rendah.Untuk spesifisitas maksimum, campuran RNA/primer dapat ditransfer langsung dari suhu denaturasi 65°C ke suhu inkubasi transkripsi terbalik dan ditambahkan ke campuran reaksi 2× yang telah dihangatkan sebelumnya (start panas sintesis cDNA).Ini membantu mencegah pasangan basa antarmolekul pada suhu rendah.Beberapa transisi temperatur yang diperlukan untuk RT-PCR dapat disederhanakan dengan menggunakan thermal cycler.
3. Mengurangi kontaminasi DNA genom:
Kesulitan potensial yang dihadapi dengan RT-PCR adalah kontaminasi DNA genom pada RNA.Penggunaan metode isolasi RNA yang baik, seperti Reagen Trizol, akan mengurangi jumlah DNA genom yang mengkontaminasi sediaan RNA.Untuk menghindari produk yang berasal dari DNA genomik, RNA dapat diobati dengan DNase I tingkat amplifikasi untuk menghilangkan DNA yang terkontaminasi sebelum transkripsi terbalik.Pencernaan DNase I diakhiri dengan menginkubasi sampel dalam 2,0 mM EDTA selama 10 menit pada suhu 65°C.EDTA dapat mengkelat ion magnesium, mencegah hidrolisis RNA yang bergantung pada ion magnesium pada suhu tinggi.
Untuk memisahkan cDNA yang diamplifikasi dari produk amplifikasi DNA genomik yang terkontaminasi, primer dapat dirancang agar setiap anil untuk memisahkan ekson.Produk PCR yang berasal dari cDNA akan lebih pendek daripada yang berasal dari DNA genom yang terkontaminasi.Selain itu, percobaan kontrol tanpa transkripsi terbalik dilakukan pada setiap templat RNA untuk menentukan apakah fragmen tertentu berasal dari DNA genomik atau cDNA.Produk PCR yang diperoleh tanpa transkripsi balik berasal dari genom.
Waktu posting: 16 Mei-2023








