Spesifisitas deteksi
Dalam kebanyakan kasus, tujuan desain primer adalah untuk memaksimalkan spesifisitas PCR.Ini ditentukan oleh pengaruh banyak variabel yang kurang lebih dapat diprediksi.Salah satu variabel penting adalah urutan pada ujung 3' primer.
Yang penting, uji PCR yang dirancang untuk spesifisitas lebih cenderung mempertahankan efisiensi tinggi pada rentang dinamis yang luas, karena uji tersebut tidak menghasilkan produk amplifikasi non-spesifik, sehingga bersaing dengan reagen PCR atau menghambat reaksi amplifikasi utama.
Tentu saja, dalam beberapa kasus, spesifisitas bukanlah yang paling penting, misalnya, ketika tujuannya adalah untuk mengukur patogen yang terkait erat tetapi berbeda, desain khusus, standar pengoptimalan dan verifikasi diperlukan.
Kurva peleburan adalah metode standar untuk menilai spesifisitas amplikon, setidaknya dalam hal apakah akan memperkuat satu target.Namun, harus ditekankan bahwa kurva leleh dapat menyesatkan karena, misalnya, dapat dipengaruhi oleh efek gabungan dari primer suboptimal dan konsentrasi templat yang rendah.
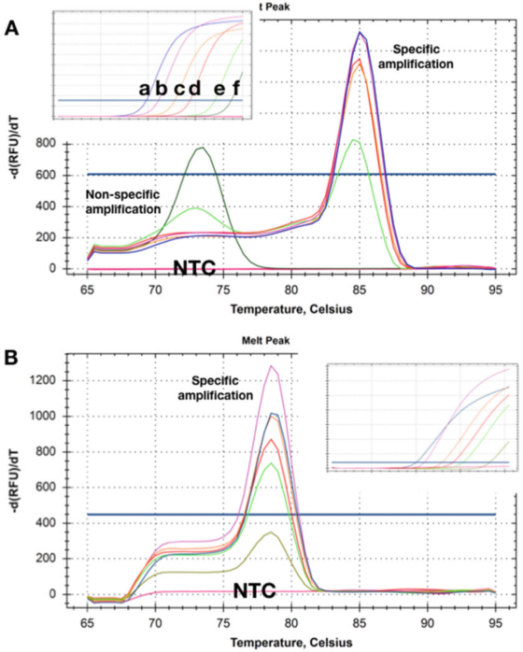
P5 |Kurva leleh menunjukkan pergeseran Tm yang diperoleh dari dua deteksi jumlah yang berbeda dari dua DNA target.
A. Pada konsentrasi yang lebih tinggi (ad)), tidak ada dimer primer yang jelas setelah pengukuran qPCR selesai.Ketika konsentrasi templat berkurang hingga 50 salinan (e), produk non-spesifik mulai muncul dan menjadi satu-satunya produk pada konsentrasi terendah (f).
B. Tes mencatat Tms yang sama pada semua konsentrasi target, dan tidak ada dimer primer yang jelas bahkan pada konsentrasi terendah (5 salinan).Saat menggunakan kedua metode deteksi ini, tidak ada produk amplifikasi yang terdeteksi di NTC.
P5 menunjukkan kurva disolusi yang diperoleh dengan sampel di mana template hadir pada konsentrasi yang berbeda.P 5a menunjukkan bahwa pada dua konsentrasi terendah, Tms dari produk amplifikasi non-spesifik yang dihasilkan lebih rendah daripada amplikon spesifik.
Jelas, metode deteksi ini tidak dapat diandalkan untuk mendeteksi target yang ada dalam konsentrasi rendah.
Menariknya, NTC, yaitu sampel tanpa DNA sama sekali, tidak merekam produk amplifikasi (non-spesifik), yang menunjukkan bahwa DNA genom latar belakang dapat berpartisipasi dalam amplifikasi/polimerisasi non-spesifik.
Kadang-kadang primer latar belakang dan amplifikasi non-spesifik seperti itu tidak dapat diperbaiki, tetapi seringkali memungkinkan untuk merancang metode deteksi yang tidak memiliki amplifikasi non-spesifik dalam konsentrasi templat dan NTC (P 5b) apa pun.
Di sini, bahkan merekam amplifikasi konsentrasi target dengan Cq 35 akan menghasilkan kurva disolusi tertentu.Demikian pula, NTC tidak menunjukkan tanda-tanda amplifikasi non-spesifik.Kadang-kadang, perilaku deteksi mungkin bergantung pada larutan induk, dan hanya amplifikasi non-spesifik yang terdeteksi pada komposisi buffer tertentu, yang mungkin terkait dengan konsentrasi Mg2+ yang berbeda.
Stabilitas deteksi
Pengoptimalan Ta adalah langkah yang berguna dalam verifikasi empiris dan proses pengoptimalan deteksi qPCR.Ini memberikan indikasi langsung kekokohan set primer dengan menunjukkan suhu (atau kisaran suhu) yang menghasilkan Cq terendah tanpa memperkuat NTC.
Perbedaan sensitivitas dua hingga empat kali lipat mungkin tidak penting bagi orang dengan ekspresi mRNA tinggi, tetapi untuk tes diagnostik, ini mungkin berarti perbedaan antara hasil positif dan negatif palsu.
Properti Ta dari primer qPCR dapat sangat bervariasi.Beberapa tes tidak terlalu kuat, dan jika tidak dilakukan di bawah nilai Ta optimal dari primer, mereka akan cepat runtuh.
Ini penting karena jenis pendeteksian ini sering bermasalah di dunia nyata, dan kemurnian sampel, konsentrasi DNA, atau keberadaan DNA lain mungkin tidak optimal.
Selain itu, jumlah salinan target dapat bervariasi dalam rentang yang luas, dan reagen, peralatan plastik, atau instrumen mungkin berbeda dari yang digunakan saat menyiapkan pengujian.
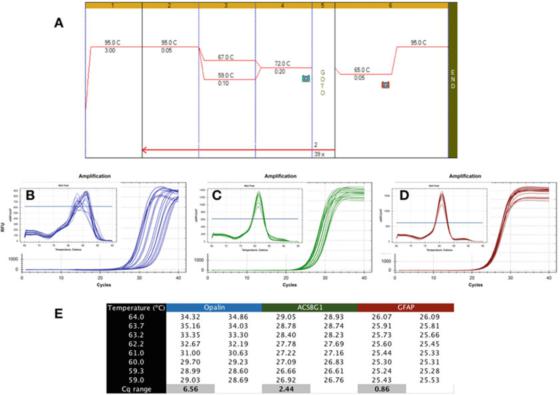
P6|Gradien suhu menunjukkan ketahanan deteksi PCR yang berbeda.
A. Gunakan mastermix Sensifast SYBR Bioline (nomor katalog BIO-98050) untuk melakukan PCR pada cDNA yang dibuat dari RNA otak manusia.
B. Gunakan instrumen CFX qPCR Bio-Rad untuk merekam peta amplifikasi dan kurva disolusi apalene (NM_033207, F: GCCATGGAGGAAAGTGACAGACC, R: CTCATGTGTGGGTGATCTCCTAGG).
C. Grafik amplifikasi dan kurva leleh ACSBG1 (NM_015162.4, F: CTACACTTCCGGCACCACTGG, R: GTCCACGTGATATTGTCTTGACTCAG).
D. Grafik amplifikasi dan kurva disolusi GFAP (NM_002055.5, F: TGGAGAGGAAGATTGAGTCGCTGG, R: CGAACCTCCTCCTCGTGGATCTTC).
E. Cqs direkam pada suhu anil yang berbeda, menunjukkan perbedaan Cq yang direkam di bawah gradien suhu 7C.
P 6 menunjukkan hasil khas dari tes yang tidak diinginkan, di mana qPCR dilakukan menggunakan Tas gradien antara 59C dan 67C (P 6a), menggunakan primer untuk tiga gen khusus otak manusia.
Dapat dilihat dari grafik amplifikasi bahwa primer Opalin jauh dari ideal karena kisaran Ta optimalnya sangat sempit (Gambar 6b), yaitu Cqs tersebar luas, menghasilkan Cqs secara signifikan dibandingkan dengan Cqs optimalnya Rendah.
Metode deteksi ini tidak stabil dan dapat menyebabkan amplifikasi suboptimal.Oleh karena itu, pasangan primer ini harus didesain ulang.Selain itu, analisis kurva leleh (inset) menunjukkan bahwa spesifisitas metode deteksi ini juga dapat menimbulkan masalah, karena kurva leleh setiap Ta berbeda.
Metode deteksi ACSBG1 yang ditunjukkan pada P 6c lebih kuat daripada metode deteksi Opalin di atas, tetapi masih jauh dari ideal, dan kemungkinan dapat ditingkatkan.
Namun, kami menekankan bahwa tidak diperlukan hubungan antara ketahanan dan spesifisitas, karena kurva disolusi yang dihasilkan oleh metode deteksi ini menunjukkan nilai puncak yang sama di semua Tas (inset).
Di sisi lain, uji ketangguhan jauh lebih toleran, menghasilkan Cq serupa di berbagai Tas, seperti dalam uji GFAP yang ditunjukkan pada P 6d.
Perbedaan Cqs yang diperoleh pada kisaran 8 derajat Celcius yang sama kurang dari 1, dan kurva disolusi (inset) menegaskan karakteristik deteksi pada kisaran suhu ini.Perlu dicatat bahwa Tas yang dihitung dan kisaran Ta yang sebenarnya mungkin sangat berbeda.
Ada banyak pedoman yang dirancang untuk membantu para peneliti merancang primer yang efisien, sebagian besar didasarkan pada aturan yang telah lama ada dan banyak perhatian telah diberikan pada ujung ke-3 dari primer.Sering disarankan untuk memasukkan G atau C pada ujung 3' dan dua basis G atau C (penjepit GC), tetapi tidak lebih dari dua dari 5 basis terakhir.
Dalam praktiknya, aturan ini dapat memandu peneliti, tetapi belum tentu benar dalam semua keadaan.
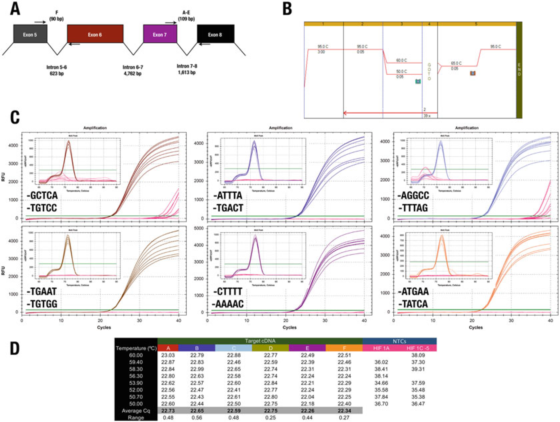
P7 |Ujung 3 dari primer memiliki sedikit efek pada spesifisitas atau efisiensi.
A. Posisi primer untuk gen manusia HIF-1α (NM_181054.2).
B. Gunakan cairan induk Agilent Brilliant III SYBR Green (Kat. No. 600882) untuk memperkuat enam item tes.
C. Grafik amplifikasi dan kurva leleh yang direkam oleh instrumen CFX qPCR Bio-Rad dan primer ujung 3.NTC ditampilkan dengan warna merah.
D. Catatan Cqs dari setiap item tes
Misalnya, hasil di P 7 bertentangan dengan aturan ujung 3.Semua desain pada dasarnya menghasilkan hasil yang sama, dengan hanya dua kombinasi primer yang mengarah ke amplifikasi non-spesifik di NTC.
Namun, kami tidak dapat mendukung efek klip GC, karena dalam hal ini, menggunakan A atau T sebagai maksimal 30 basis tidak mengurangi spesifisitas.
Uji C, di mana primer F berakhir di GGCC, mencatat Cq di NTC, menunjukkan bahwa seseorang mungkin ingin menghindari urutan ini pada akhir ke-30.Kami menekankan bahwa satu-satunya cara untuk menentukan urutan 3′end terbaik dari pasangan primer adalah dengan mengevaluasi beberapa kandidat primer secara eksperimental.
Efisiensi amplifikasi
Yang penting, meskipun deteksi PCR non-spesifik tidak pernah menjadi spesifik, efisiensi amplifikasi dapat disesuaikan dan dimaksimalkan dengan berbagai cara dengan mengubah kondisi enzim, cairan induk, aditif, dan siklus.
Untuk mengevaluasi efisiensi deteksi PCR, yang terbaik adalah menggunakan pengenceran serial 10 atau 5 kali asam nukleat target, yaitu “metode kurva standar”.
Jika amplikon PCR atau target DNA sintetik digunakan untuk menghasilkan kurva standar, pengenceran serial dari target ini harus dicampur dengan jumlah DNA latar yang konstan (seperti DNA genomik).
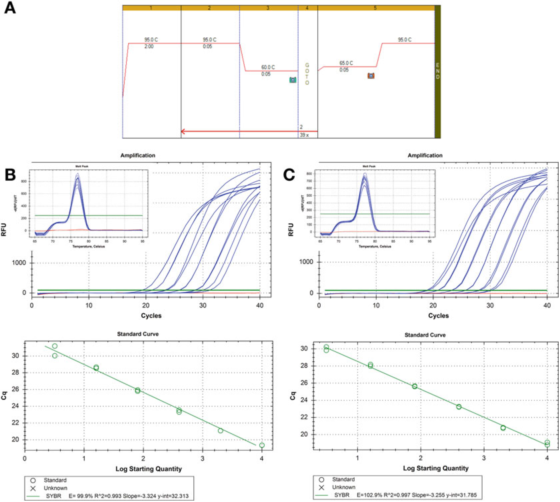
P8 |Kurva dilusi untuk mengevaluasi efisiensi PCR.
A. Gunakan primer untuk HIF-1: F: AAGAACTTTTAGGCCGCTCA dan R: TGTCCTGTGGTGACTTGTCC dan mastermix Agilent's Brilliant III SYBR Green (nomor katalog 600882) untuk PCR dan kondisi kurva leleh.
B. 100 ng RNA ditranskripsi terbalik, diencerkan 2 kali, dan sampel cDNA yang diencerkan secara serial diencerkan 5 kali menjadi 1 ng DNA genomik manusia.Kurva leleh ditunjukkan pada inset.
C. Reaksi RT, pengenceran, dan pengenceran serial diulangi untuk sampel cDNA kedua, dan hasilnya serupa.
P 8 menunjukkan dua kurva standar, menggunakan metode deteksi yang sama pada dua sampel cDNA yang berbeda, hasilnya adalah efisiensi yang sama, sekitar 100%, dan nilai R2 juga serupa, yaitu tingkat kecocokan antara data eksperimen dan garis regresi atau derajat linearitas data.
Kedua kurva standar dapat dibandingkan, tetapi tidak persis sama.Jika tujuannya adalah untuk mengukur target secara akurat, harus dicatat bahwa memberikan salinan perhitungan angka tanpa menjelaskan ketidakpastian tidak dapat diterima.
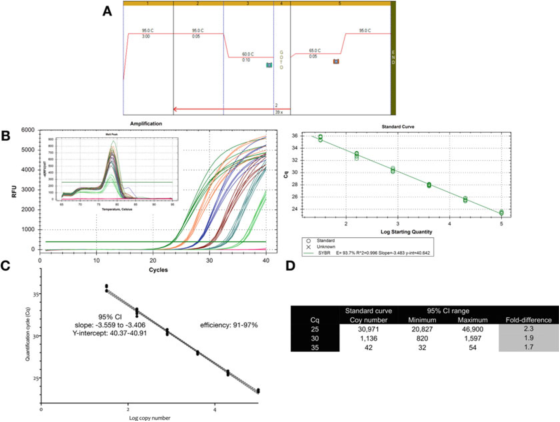
P9 |Ketidakpastian pengukuran terkait dengan kuantifikasi menggunakan kurva standar.
A. Gunakan primer untuk GAPDH (NM_002046) untuk melakukan PCR dan kondisi kurva leleh.F: ACAGTTGCCATGTAGACC dan R: TAACTGGTTGAGCACAGG dan mastermix Sensifast SYBR Bioline (nomor katalog BIO-98050).
B. Bagan amplifikasi, kurva leleh, dan kurva standar yang direkam dengan instrumen CFX qPCR Bio-Rad.
C. Grafik kurva standar dan interval kepercayaan (CI) 95%.
D. Jumlah salinan dan interval kepercayaan 95% dari ketiga nilai Cq yang diperoleh dari kurva pengenceran.
P 9 menunjukkan bahwa untuk pengujian yang dioptimalkan, variabilitas inheren dari kurva standar tunggal kira-kira 2 kali (interval kepercayaan 95%, minimum hingga maksimum), yang mungkin merupakan variabilitas terkecil yang dapat diharapkan.
Produk terkait:
Kit PCR Langsung Jaringan Hewan
Waktu posting: Sep-30-2021